디지털 슬라이드 스캐너와 NEGCUT 결합한 플랫폼으로 최대 100장 동시 처리·진단 정확도 95.9%…“임상 적용 기대”

성균관대학교 생명물리학과 박별리 교수 연구팀과 삼성서울병원 김형경 교수 연구팀이 화학적 염색 없이도 병리 진단에 필요한 H&E 등가 이미지를 자동 생성하고 암을 분류할 수 있는 고처리량 AI 기반 가상염색 기술을 개발했다.
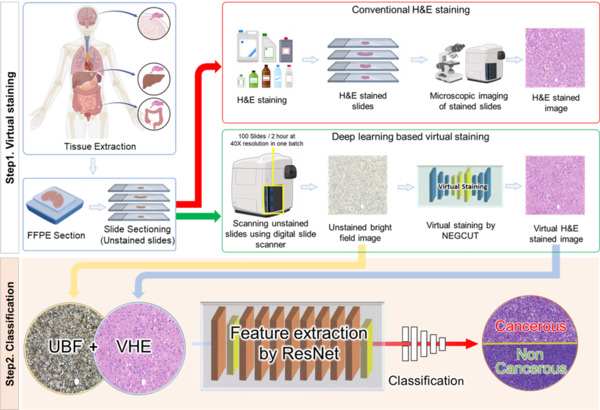
H&E(hematoxylin & eosin) 염색은 핵과 조직 구조를 선명하게 보여주는 병리 검사의 기본 절차지만, 화학 처리와 숙련 인력이 필요하고 한 장의 슬라이드를 준비하는 데 통상 20~30분이 소요되는 등 시간과 비용 부담이 크다. 이러한 이유로 대규모 임상 워크플로우에 적용하기 어려운 한계가 존재했다.
연구팀은 최대 100장의 슬라이드를 동시에 스캔할 수 있는 고해상도 디지털 슬라이드 스캐너와 자체 개발한 NEGCUT 딥러닝 모델을 결합한 플랫폼을 구축했다.
이 플랫폼은 염색되지 않은 명시야(unstained bright-field) 영상을 입력으로 받아 진단 수준의 가상 H&E 영상을 자동으로 생성해, 실제 염색 없이도 병리 분석이 가능한 ‘비염색 디지털 병리학’을 구현한다.
제안된 NEGCUT 기반 가상염색은 구조 재현성과 색 재현성에서 기존 CycleGAN·CUT·E-CUT 계열 모델보다 우수한 성능을 보였고, 무염색 영상과 가상 H&E 영상을 함께 사용하는 이중 브랜치 ResNet 기반 암 분류 모델은 95.9%의 진단 정확도를 기록했다.
또한 병리 전문의 3명이 참여한 블라인드 평가에서 가상염색 이미지가 실제 H&E와 구별하기 어려웠다는 판단을 받았다.
해당 기술은 림프절·뇌·간·갑상선·뼈 등 다양한 조직에서 안정적으로 작동했으며, 화학 염색을 생략함으로써 비용과 시간을 절감하고 조직 손상을 피할 수 있어 면역조직화학(IHC) 및 분자 분석 등 후속 연구에도 원본 샘플을 온전히 활용할 수 있다는 장점을 가진다.
박별리 교수는 이번 연구가 기존 가상염색 기술의 저속 처리 문제를 극복하고 병원에서 실제 사용하는 대량 디지털 스캔 환경에 적용 가능한 자동화 기술을 구현했다는 점에서 의의가 있다고 설명했다. 김형경 교수는 가상염색 이미지 품질이 실제 염색과 유사해 임상 적용 가능성을 앞당겼다고 평가했다.
이 연구는 한국연구재단의 여러 지원 사업과 BK21FOUR 프로젝트의 지원을 받아 수행됐으며, 연구 결과는 국제학술지 Medical Image Analysis에 게재가 확정됐다(논문명: Deep learning based label-free virtual staining and classification of human tissues using digital slide scanner).

